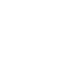構造生物学研究室
概要
分子細胞生物学的に重要な機能を有するタンパク質や核酸およびその集合体(生体超分子複合体)の立体構造を、X線や中性子線をプローブとした結晶解析や溶液散乱によって決定し、生体高分子間の多元的な相互作用に基づく機能発現の仕組みを解明する。研究対象は、細胞核内において形成されるヌクレオソーム及びその集合体、ヒストンの化学修飾やDNAメチル化を触媒する酵素、それらの認識と継承に関わるタンパク質、細胞表面受容体を初めとする細胞膜上でのシグナル伝達に関わるタンパク質などである。
スタッフ
教授 佐藤 衛(さとう まもる)

<略歴>
大阪大学大学院工学研究科博士後期課程修了(1983年)、工学博士。摂南大学薬学部研究職員・助手、大阪大学蛋白質研究所教務職員・助手・助教授を経て、1996年4月横浜市立大学大学院総合理学研究科教授、2005年4月同国際総合科学研究科教授、2009年4月同生命ナノシステム科学研究科教授、2013年4月同生命医科学研究科教授
<メッセージ>
変化の激しい世の中で、「独創的で、オンリーワンの研究を!!・・・」と肩肘はって気張らなくても、変化を楽しむ余裕さえあれば、結果は自ずと独創的になっていきます。さあ、みなさん、私たちといっしょに研究を楽しんでみませんか。ひょっとしたら独創的な成果が生まれるかもしれませんよ
大阪大学大学院工学研究科博士後期課程修了(1983年)、工学博士。摂南大学薬学部研究職員・助手、大阪大学蛋白質研究所教務職員・助手・助教授を経て、1996年4月横浜市立大学大学院総合理学研究科教授、2005年4月同国際総合科学研究科教授、2009年4月同生命ナノシステム科学研究科教授、2013年4月同生命医科学研究科教授
<メッセージ>
変化の激しい世の中で、「独創的で、オンリーワンの研究を!!・・・」と肩肘はって気張らなくても、変化を楽しむ余裕さえあれば、結果は自ずと独創的になっていきます。さあ、みなさん、私たちといっしょに研究を楽しんでみませんか。ひょっとしたら独創的な成果が生まれるかもしれませんよ
准教授 禾 晃和(のぎ てるかず)

<略歴>
京都大学大学院理学研究科化学専攻博士課程修了(2001年)理学博士。2001年高エネ研物構研・博士研究員。2002年マックスプランク生物物理研・博士研究員。2004年阪大蛋白研・助手(2007年助教)。2011年4月から横浜市立大学准教授。
<メッセージ>
構造生物学は、生物学、化学、物理学など、色々なバックグラウンドを持った人間がそれぞれの得意分野を生かして研究に取り組むことができる領域です。我々のグループでは、タンパク質分子が外界からの情報を受け取り、細胞の中へと受け渡している姿を結晶や溶液の中で再現し、原子・分子のレベルで可視化することを目指して研究を進めています。
京都大学大学院理学研究科化学専攻博士課程修了(2001年)理学博士。2001年高エネ研物構研・博士研究員。2002年マックスプランク生物物理研・博士研究員。2004年阪大蛋白研・助手(2007年助教)。2011年4月から横浜市立大学准教授。
<メッセージ>
構造生物学は、生物学、化学、物理学など、色々なバックグラウンドを持った人間がそれぞれの得意分野を生かして研究に取り組むことができる領域です。我々のグループでは、タンパク質分子が外界からの情報を受け取り、細胞の中へと受け渡している姿を結晶や溶液の中で再現し、原子・分子のレベルで可視化することを目指して研究を進めています。
准教授 有田 恭平(ありた きょうへい)

<略歴>
横浜市立大学大学院総合理学研究科博士後期課程修了(2006年)理学博士。2005年日本学術振興会特別研究員、2010年から京都大学工学研究科助教。2013年4月から横浜市立大学大学院生命医科学研究科准教授
<メッセージ>
真核生物のクロマチン構造の制御に関与するDNAメチル化やヒストン修飾の形成・認識・継承に関与するタンパク質に焦点をあて、構造生物学な観点からクロマチンの制御機構の解明を目指します。X線結晶構造解析を主体にして、NMRや高速AFMなどの構造生物学的な手法や生化学、物理化学的解析を行い、多角的に研究を進めいていきます。
横浜市立大学大学院総合理学研究科博士後期課程修了(2006年)理学博士。2005年日本学術振興会特別研究員、2010年から京都大学工学研究科助教。2013年4月から横浜市立大学大学院生命医科学研究科准教授
<メッセージ>
真核生物のクロマチン構造の制御に関与するDNAメチル化やヒストン修飾の形成・認識・継承に関与するタンパク質に焦点をあて、構造生物学な観点からクロマチンの制御機構の解明を目指します。X線結晶構造解析を主体にして、NMRや高速AFMなどの構造生物学的な手法や生化学、物理化学的解析を行い、多角的に研究を進めいていきます。
研究内容
(佐藤グループ)細胞核内におけるヌクレオソーム及びヌクレオソーム集合体の構造動態解析を行うために必要なX線及び中性子溶液散乱法の開発を行うとともに、近年タンパク質の構造・機能研究の新しいターゲットとして注目されるようになった天然変性タンパク質(Intrinsically Disordered Protein)の動的挙動を原子レベルで解析する手法の開発も行っている。

(禾グループ)細胞膜上でシグナル伝達の中でも、特に1回膜貫通型タンパク質を介したシグナル伝達を取り上げ、細胞外からのシグナルの受容と膜を隔てた細胞内へのシグナル伝達の仕組みを分子・原子のレベルで理解することを目的として構造研究を進める。主な研究対象としては、脳神経の発生に関わるシグナル伝達や膜内配列切断を介したシグナル伝達を取り上げる。1回膜貫通型の受容体やシグナル分子が、細胞膜という二次元の反応場において、弱い相互作用ながらも精巧な分子認識を行い、ダイナミックな構造変化を起こしてシグナルを変換する様子を、X線結晶解析を基盤とした構造生物学的手法によって捉えていく。
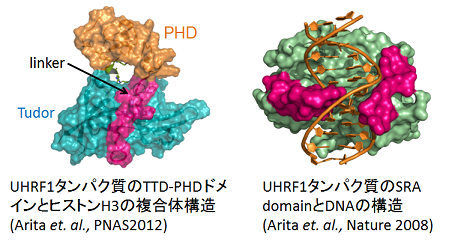
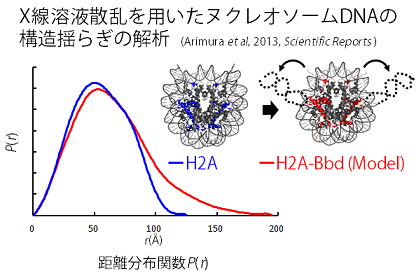
(有田グループ) 真核生物のDNAはヒストンタンパク質に巻き付きヌクレオソーム構造を基本単位としたクロマチン構造を形成する。クロマチン構造の制御にはDNAメチル化やヒストン修飾などのエピジェネティクスが関与する。エピジェネティックな情報の形成機構・認識機構・継承機構に関わる生体分子の構造生物学的な研究を行い、エピジェネティクスによるクロマチン構造の制御機構の解明を目指します。対象とする生体分子はマルチドメインタンパク質であったり超分子複合体であるので、X線結晶構造解析による高分解能かつ静的な構造情報に加えて、X線小角散乱やNMRなど動的な構造情報を組み合わせることにより、多角的に構造と機能の相関を解明していきます。
主要文献(Selected Publications)
Y. Hizukuri, T. Oda, S. Tabata, K. Tamura-Kawakami, R. Oi, M. Sato, J. Takagi, Y. Akiyama and T. Nogi A Structure-Based Model of Substrate Discrimination by a Noncanonical PDZ Tandem in the Intramembrane-Cleaving Protease RseP, Structure 22, 326 (2014).
Nishiyama A, Yamaguchi L, Sharif J, Johmura Y, Kawamura T, Nakanishi K, Shimamura S, Arita K, Kodama T, Ishikawa F, Koseki H, and Nakanishi M . Uhrf1-dependent ubiquitylation of histone H3 at lysine 23 couples maintenance DNA methylation and DNA replication. Nature 502, 249-253, 2013
J. Trewhella, W. A. Hendrickson, G. J. Kleywegt, A. Sali, M. Sato, T. Schwede, D. I. Svergun, J. A. Tainer, J. Westbrook, and H. M. Berman, Report of the wwPDB Small-Angle Scattering Task Force: Data Requirements for Biomolecular Modeling and the PDB, Structure, 21, 875-881 (2013)
K. Arita, S. Isogai, T. Oda, M. Unoki, K. Sugita, N. Sekiyama, K. Kuwata, R. Hamamoto, H. Tochio, M. Sato, M. Ariyoshi, M. Shirakawa. Recognition of modification status on a histone H3 tail by linked histone reader modules of the epigenetic regulator UHRF1 Proc Natl Acad Sci U S A. 109, 12950-12955, 2012.
H. Tachiwana, W. Kagawa, T. Shiga, A. Osakabe, Y. Miya, K. Saito, Y. Hayashi-Takanaka, T. Oda, M. Sato, S.-Y. Park, H. Kimura, and H. Kurumizaka, Crystal structure of the human centromeric nucleosome containing CENP-A, Nature 476, 232-235 (2011)
T. Nogi, N. Yasui, E. Mihara, Y. Matsunaga, M. Noda, N. Yamashita, T. Toyofuku, S. Uchiyama, Y. Goshima, A. Kumanogoh and J. Takagi Structural basis for semaphorin signalling through the plexin receptor, Nature, 467, 1123 (2010).
Nishiyama A, Yamaguchi L, Sharif J, Johmura Y, Kawamura T, Nakanishi K, Shimamura S, Arita K, Kodama T, Ishikawa F, Koseki H, and Nakanishi M . Uhrf1-dependent ubiquitylation of histone H3 at lysine 23 couples maintenance DNA methylation and DNA replication. Nature 502, 249-253, 2013
J. Trewhella, W. A. Hendrickson, G. J. Kleywegt, A. Sali, M. Sato, T. Schwede, D. I. Svergun, J. A. Tainer, J. Westbrook, and H. M. Berman, Report of the wwPDB Small-Angle Scattering Task Force: Data Requirements for Biomolecular Modeling and the PDB, Structure, 21, 875-881 (2013)
K. Arita, S. Isogai, T. Oda, M. Unoki, K. Sugita, N. Sekiyama, K. Kuwata, R. Hamamoto, H. Tochio, M. Sato, M. Ariyoshi, M. Shirakawa. Recognition of modification status on a histone H3 tail by linked histone reader modules of the epigenetic regulator UHRF1 Proc Natl Acad Sci U S A. 109, 12950-12955, 2012.
H. Tachiwana, W. Kagawa, T. Shiga, A. Osakabe, Y. Miya, K. Saito, Y. Hayashi-Takanaka, T. Oda, M. Sato, S.-Y. Park, H. Kimura, and H. Kurumizaka, Crystal structure of the human centromeric nucleosome containing CENP-A, Nature 476, 232-235 (2011)
T. Nogi, N. Yasui, E. Mihara, Y. Matsunaga, M. Noda, N. Yamashita, T. Toyofuku, S. Uchiyama, Y. Goshima, A. Kumanogoh and J. Takagi Structural basis for semaphorin signalling through the plexin receptor, Nature, 467, 1123 (2010).
 生命医科学研究科
生命医科学研究科