生命情報科学研究室
概要
タンパク質、核酸、その複合体などの立体構造形成や機能発現のメカニズムを原子レベルで明らかにするために、スーパーコンピュータを用いた分子シミュレーションや構造バイオインフォマティクスによる研究を行っています。さらに、それらの方法をドラッグデザインに応用する計算創薬研究を行っています。
スタッフ
教授 木寺 詔紀(きでら あきのり)

<略歴>
京都大学工学研究科高分子化学専攻博士課程修了(1982年)工学博士。1996年から京都大学大学院理学研究科助教授。2001年4月から横浜市立大学大学院教授。
<メッセージ>
蛋白質の機能・物性を理解するために、物理学・化学・情報科学などのあらゆる方法を使って研究を行っている。特に、データベース解析と分子シミュレーションを相補的・融合的に用いた研究を行うことによって、分子機能を超えた高次機能に迫る可能性を模索したいと考えている。
京都大学工学研究科高分子化学専攻博士課程修了(1982年)工学博士。1996年から京都大学大学院理学研究科助教授。2001年4月から横浜市立大学大学院教授。
<メッセージ>
蛋白質の機能・物性を理解するために、物理学・化学・情報科学などのあらゆる方法を使って研究を行っている。特に、データベース解析と分子シミュレーションを相補的・融合的に用いた研究を行うことによって、分子機能を超えた高次機能に迫る可能性を模索したいと考えている。
教授 池口 満徳(いけぐち みつのり)

<略歴>
東京大学大学院農学系研究科応用生命工学専攻博士課程修了(1994年)博士(農学)。1996年から東京大学大学院農学生命科学研究科助手。2001年4月から横浜市立大学大学院助教授。2007年より同大学院准教授。
<メッセージ>
タンパク質の機能発現やリガンド結合のメカニズムを分子動力学シミュレーションを主に用いて研究を進めています。製薬企業との連携やスーパーコンピュータ ポスト「京」のプロジェクトにおいて、計算創薬研究を推進しています。
東京大学大学院農学系研究科応用生命工学専攻博士課程修了(1994年)博士(農学)。1996年から東京大学大学院農学生命科学研究科助手。2001年4月から横浜市立大学大学院助教授。2007年より同大学院准教授。
<メッセージ>
タンパク質の機能発現やリガンド結合のメカニズムを分子動力学シミュレーションを主に用いて研究を進めています。製薬企業との連携やスーパーコンピュータ ポスト「京」のプロジェクトにおいて、計算創薬研究を推進しています。
助教 渕上 壮太郎(ふちがみ そうたろう)

<略歴>
東京大学大学院総合文化研究科広域科学専攻博士課程修了(2002年)、博士(学術)。科学技術振興事業団・計算科学技術研究員を経て、2004年4月から横浜市立大学大学院助手。2007年4月より同大学院助教。
<メッセージ>
タンパク質は生命活動に必要な様々な機能を実現していますが、その実現にはタンパク質に特有な「ダイナミクス」が重要な役割を果たしています。このダイナミクスの原子レベルでの理解と、背後に潜む一般原理の発見・解明を目指し、コンピュータ・シミュレーションを用いて研究を行なっています。
東京大学大学院総合文化研究科広域科学専攻博士課程修了(2002年)、博士(学術)。科学技術振興事業団・計算科学技術研究員を経て、2004年4月から横浜市立大学大学院助手。2007年4月より同大学院助教。
<メッセージ>
タンパク質は生命活動に必要な様々な機能を実現していますが、その実現にはタンパク質に特有な「ダイナミクス」が重要な役割を果たしています。このダイナミクスの原子レベルでの理解と、背後に潜む一般原理の発見・解明を目指し、コンピュータ・シミュレーションを用いて研究を行なっています。
研究内容
生命現象を分子レベルで理解するため、タンパク質や核酸(DNA, RNA)といった生体高分子の働き(機能)の研究が進展してきました。これら生体高分子の働きは、分子の運動として理解可能です。その研究方法には、バイオインフォマティクスの立場から、構造既知のタンパク質に構造-機能相関の経験的ルールを探る研究と、分子動力学シミュレーションの立場から、原子間相互作用に基づいた解析を行うという研究があります。我々は、これらふたつの方法を駆使することにより、生体高分子の機能発現メカニズムを分子レベルで解明することを目指しています。
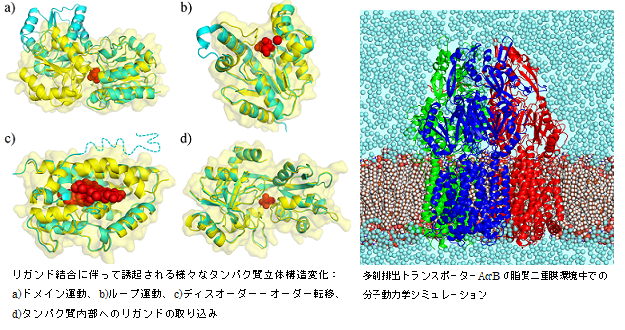
バイオインフォマティクスの研究としては、低分子リガンドの結合に伴うタンパク質立体構造変化のデータベース構築を挙げることができます。この研究では、図に示したようなタンパク質の様々な立体構造変化を網羅的かつ系統的に分類するとともに、立体構造変化の因果関係について解析し、注釈付けを行いました。このようにタンパク質立体構造変化の全体像を把握・理解することで、タンパク質機能の一般論を展開することが可能となります。
分子シミュレーションのターゲットとして最も注目しているのが膜タンパク質です。多くの医薬品の標的タンパク質も膜タンパク質です。これまでの成果のひとつとして、右下図に示した多剤排出トランスポーターAcrBの研究があります。多剤排出トランスポーターは、薬が効かなくなる薬剤耐性の原因でもあり、その薬剤排出メカニズムの解明が重要とされています。さらに、製薬企業とも連携しつつ、分子シミュレーションを創薬に応用するin silicoドラッグデザイン研究も展開しています。
バイオインフォマティクスの研究としては、低分子リガンドの結合に伴うタンパク質立体構造変化のデータベース構築を挙げることができます。この研究では、図に示したようなタンパク質の様々な立体構造変化を網羅的かつ系統的に分類するとともに、立体構造変化の因果関係について解析し、注釈付けを行いました。このようにタンパク質立体構造変化の全体像を把握・理解することで、タンパク質機能の一般論を展開することが可能となります。
分子シミュレーションのターゲットとして最も注目しているのが膜タンパク質です。多くの医薬品の標的タンパク質も膜タンパク質です。これまでの成果のひとつとして、右下図に示した多剤排出トランスポーターAcrBの研究があります。多剤排出トランスポーターは、薬が効かなくなる薬剤耐性の原因でもあり、その薬剤排出メカニズムの解明が重要とされています。さらに、製薬企業とも連携しつつ、分子シミュレーションを創薬に応用するin silicoドラッグデザイン研究も展開しています。
主要文献(Selected Publications)
R. Koike, M. Ota, A. Kidera: Hierarchical description and extensive classification of protein structural changes by Motion Tree. J. Mol. Biol., 426, 752-762 (2014).
Y. Ida, A. Kidera: The conserved Arg241-Glu439 salt bridge determines flexibility of the inositol 1,4,5-trisphosphate receptor binding core in the ligand-free state. Proteins, 81, 1699-1708 (2013)
Yamane, S. Murakami, M. Ikeguchi: Functional rotation induced by alternating protonation states in the multidrug transporter AcrB: All-atom molecular dynamics simulations. Biochemistry, 52, 7648-7658 (2013).
Y. Ito, M. Ikeguchi: Mechanism of the αβ conformational change in F1-ATPase after ATP hydrolysis: free energy simulations. Biophys. J., 108, 85-97, (2015).
Y. Naritomi, S. Fuchigami: Slow dynamics of a protein backbone in molecular dynamics simulation revealed by time-structure based independent component analysis. J. Chem. Phys., 139, 215102 (2013).
Y. Ida, A. Kidera: The conserved Arg241-Glu439 salt bridge determines flexibility of the inositol 1,4,5-trisphosphate receptor binding core in the ligand-free state. Proteins, 81, 1699-1708 (2013)
Yamane, S. Murakami, M. Ikeguchi: Functional rotation induced by alternating protonation states in the multidrug transporter AcrB: All-atom molecular dynamics simulations. Biochemistry, 52, 7648-7658 (2013).
Y. Ito, M. Ikeguchi: Mechanism of the αβ conformational change in F1-ATPase after ATP hydrolysis: free energy simulations. Biophys. J., 108, 85-97, (2015).
Y. Naritomi, S. Fuchigami: Slow dynamics of a protein backbone in molecular dynamics simulation revealed by time-structure based independent component analysis. J. Chem. Phys., 139, 215102 (2013).
 生命医科学研究科
生命医科学研究科