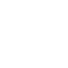機能ゲノム科学研究室
概要
転写因子やエピジェネティック修飾などを通じて制御される遺伝情報発現機構、その結果作られる非コードRNAやタンパク質の役割、更には細胞が機能を果たすために必要な細胞内ネットワークなど、細胞内で起きている 分子レベルでの事象を網羅的・体系的に理解することを目指します。次世代シーケンサーを駆使したトランスクリプトーム・エピジェネティックマーク等の網羅的測定技術や、大量データを効率的に解析するゲノム情報的解析技術、遺伝子発現を人為的に操作する技術、等を駆使する他、これらの技術開発も行います。
スタッフ
大学院客員教授 粕川 雄也 (かすかわ たけや) ResearchMap →

<略歴>
大阪大学大学院基礎工学研究科 博士(情報科学)2005年 大阪大学 助手、NTTソフトウェア、国立研究法人理化学研究所 研究員、専門職研究員、ユニットリーダーを経て、現生命医科学研究センター、チームリーダー
<メッセージ>
シーケンサーなどゲノム解析のための測定技術が大きく進歩したことで、様々な情報を測定することができるようになりました。しかし一方でゲノムから始まる生命現象の理解はまだまだ途中です。そこで、いわゆるウェットと呼ばれる実験・解析技術や、ドライと呼ばれる計算機処理・データ科学などの様々な手法を駆使してゲノム機能の理解を目指します。
大学院客員准教授 高橋 葉月(たかはし はづき)

<略歴>
製薬系ベンチャー企業を経て、2008年に理化学研究所に入所。2018年に横浜市立大学大学院生命医科学研究科にて理学の博士(論文博士)を取得。現在は理化学研究所 生命医科学研究センター トランスクリプトーム研究チームにて、特別任期制研究員として就業中。
<メッセージ>
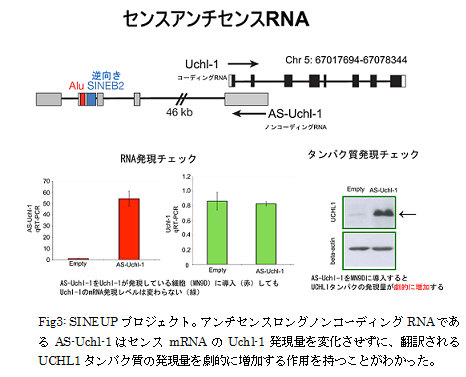
我々は、ゴミDNAのとして考えられてきたレトロトランスポゾン遺伝子の一部が、生体内で重要な役割を担っていることを発見しました。蛋白質に翻訳されない非コードRNAの中にはレトロトランスポゾンが多く発現していますが、未だその多くの機能がわかっていません。我々の研究室では、ゲノム解析から得られた情報にさらなる実験手法を組み合わせることで、レトロトランスポゾン遺伝子の機能を解明しています。
製薬系ベンチャー企業を経て、2008年に理化学研究所に入所。2018年に横浜市立大学大学院生命医科学研究科にて理学の博士(論文博士)を取得。現在は理化学研究所 生命医科学研究センター トランスクリプトーム研究チームにて、特別任期制研究員として就業中。
<メッセージ>
我々は、ゴミDNAのとして考えられてきたレトロトランスポゾン遺伝子の一部が、生体内で重要な役割を担っていることを発見しました。蛋白質に翻訳されない非コードRNAの中にはレトロトランスポゾンが多く発現していますが、未だその多くの機能がわかっていません。我々の研究室では、ゲノム解析から得られた情報にさらなる実験手法を組み合わせることで、レトロトランスポゾン遺伝子の機能を解明しています。
大学院客員助教 信定 知江 (のぶさだ ともえ)

<略歴>
総合研究大学院大学生命科学研究科 博士(理学)2004年 基礎生物学研究所 研究員、理化学研究所 研究員、科学技術振興機構バイオサイエンスデータベースセンター 研究員を経て、現在は理化学研究所生命医科学研究センター生命医科学大容量データ技術研究チーム 研究員
<メッセージ>
総合研究大学院大学生命科学研究科 博士(理学)2004年 基礎生物学研究所 研究員、理化学研究所 研究員、科学技術振興機構バイオサイエンスデータベースセンター 研究員を経て、現在は理化学研究所生命医科学研究センター生命医科学大容量データ技術研究チーム 研究員
<メッセージ>
ウェット、ドライ、という言葉を研究環境において聞くようになって久しく、お互いの壁は以前ほど高くないように感じます。当研究室には様々なバックグラウンドの研究者が密に連携をとりながら研究を進めていく環境が整っており、多様な知見をベースにいろいろな角度からゲノムの謎の解明を目指しています。
研究内容
We are interested in the information encoded in the genome, functional genomics (transcriptome and epigenome), with aiming to understand the entire system across the genome, monitor the functional signature, and ultimately manipulate cells as intended. Our unique technologies in transcriptome (full-length cDNA to determine full structure of individual genes and CAGE to genome-wide quantify transcription initiation activity) have provided important baseline in genome annotation and decoded complex architecture of transcription in vertebrates. Our interdisciplinary approach ranges from technology development in genomics and molecular biology and information science so that we can fully utilize next generation sequencer, to cell biology, hacking the code of the genome in actually living cells. The laboratory is also engaged in the study of non-protein coding RNAs, which often reveal completely novel functions and mechanisms of action. We also aim to contribute the society by modifying/reprogramming cells and engineering lncRNAs for future therapies. Please contact us if you are willing to join our research activities
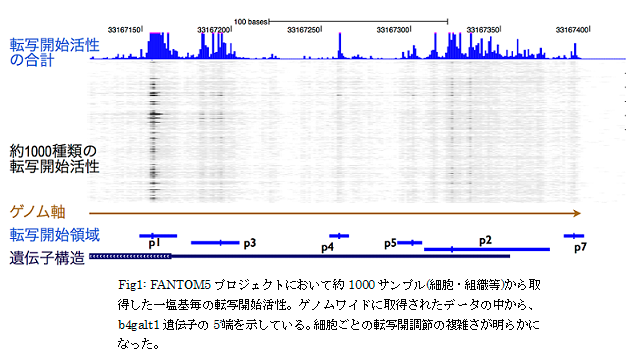
私たちはゲノムにコードされている情報を理解するために、トランスクリプトームやエピゲノム解析等の機能ゲノミクスで研究を行っています。我々独自のトランスクリプトーム解析技術(遺伝子の全体構造を捕まえる完全長cDNA技術や、転写開始活性をゲノムワイドに定量化するCAGE法)は、ゲノムを理解する上での重要な基盤として、またゲノムからの転写開始様式を理解する上で重要な役割を果たしてきました。これら技術の活用や、新技術の開発を通じてゲノムがコードする分子システムやネットワークの全体像・機能的な特徴を理解したい、また究極的には細胞を思うがままに操作できないかと考えています。私たちの研究室ではゲノム科学・情報科学や分子生物学・細胞生物学等を様々に駆使した学際的なアプローチを通じ、近年様々な生物現象への関与が分かってきたノンコーディングRNAの新規機能の解析や医療への応用、細胞の分子メカニズムに基づくダイレクトリプログラミングなどの細胞機能操にチャレンジしています。生物現象を分子レベルで理解し操作することに興味のある皆さんと研究ができることを楽しみにしています。
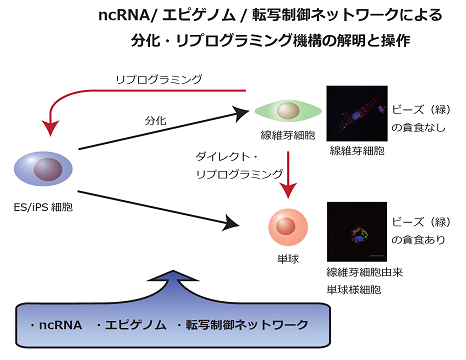
Fig2. 転写制御ネットワークの再構築による単球のダイレクダイレクト・リプログラミング
主要文献(Selected Publications)
‣Abugessaisa I, Hasegawa A, Noguchi S, Cardon M, Watanabe K, Takahashi M, Suzuki H, Katayama S, Kere J, Kasukawa T. SkewC: Identifying cells with skewed gene body coverage in single-cell RNA sequencing data. iScience. 25(2):103777 (2022). PMID: 35146392
‣Yoshino T, Suzuki T, Nagamatsu G, Yabukami H, Ikegaya M, Kishima M, Kita H, Imamura T, Nakashima K, Nishinakamura R, Tachibana M, Inoue M, Shima Y, Morohashi K, Hayashi K. Generation of ovarian follicles entirely derived from mouse pluripotent stem cells. Science 16; 373(6552)(2021) PMID: 34437124
‣Abugessaisa I, Noguchi S, Hasegawa A, Kondo A, Kawaji H, Carninci P, Kasukawa T. refTSS: A Reference Data Set for Human and Mouse Transcription Start Sites. J Mol Biol. 431(13):2407-2422 (2019). PMID: 31075273
‣Toki N, Takahashi H, Sharma H, Valentine M NZ, Rahman M F-Ur, Zucchelli S, Gustincich S, Carninci P. SINEUP long non-coding RNA acts via PTBP1 and HNRNPK to promote translational initiation assemblies. Nucleic Acids Res 48(20):11626-11644 (2020) PMID: 33130894
‣Toki N, Takahashi H, Zucchelli S, Gustincich S, Carninci P. Synthetic in vitro transcribed lncRNAs (SINEUPs) with chemical modifications enhance target mRNA translation. FEBS Letters 594(24):4357-4369 (2020) PMID: 33012004
‣Takahashi H, Sharma H, Carninci P. Cell Based Assays of SINEUP Non-coding RNAsThat Can Specifically Enhance mRNA Translation. J Vis Exp. 144 (2019) PMID: 30774120