細胞構造ダイナミクス研究室
概要
私たちの研究室では、細胞内で生体分子が働く仕組みを詳細に明らかにするために、クライオ電子顕微鏡等 最先端の技術を用いた構造生物学研究を行っています。特に、細胞膜で受け取った情報を細胞内で伝えるシグナル伝達系と、その指示を受けて遺伝子の発現を行う転写系のタンパク質群に着目した構造・機能解析を進めています。生体分子やそれらが集まって形成される様々な複合体等の構造やダイナミクスを明らかにすることで、細胞内シグナル伝達およびクロマチン内での転写の仕組みを分子のレベルで明らかにするとともに、その破綻による疾患メカニズムの理解にも迫ります。
スタッフ

<略歴>
1995年 東京大学大学院理学系研究科修了(博士(理学))
1995年 理化学研究所 研究員
2001年 理化学研究所 チームリーダー
2007年 理化学研究所 上級研究員
2013年 理化学研究所 チームリーダー
2021年 横浜市立大学大学院客員教授を兼任
<メッセージ>
細胞膜で受け取った情報を細胞内でどのように伝えて特異的な反応を引き起こすのか、そのメカニズムをタンパク質科学・構造生物学的手法で明らかにしていきたいと考えています。
1995年 東京大学大学院理学系研究科修了(博士(理学))
1995年 理化学研究所 研究員
2001年 理化学研究所 チームリーダー
2007年 理化学研究所 上級研究員
2013年 理化学研究所 チームリーダー
2021年 横浜市立大学大学院客員教授を兼任
<メッセージ>
細胞膜で受け取った情報を細胞内でどのように伝えて特異的な反応を引き起こすのか、そのメカニズムをタンパク質科学・構造生物学的手法で明らかにしていきたいと考えています。
大学院客員教授 関根 俊一(せきね しゅんいち) ResearchMap →

<略歴>
1997年 東京大学大学院理学系研究科修了(博士(理学))
1997年 日本学術振興会 特別研究員PD
1998年 理化学研究所 協力研究員
1999年 理化学研究所 基礎科学特別研究員
2001年 理化学研究所 研究員
2004年 東京大学大学院理学系研究科 講師
2010年 東京大学大学院理学系研究科 特任准教授
2012年 理化学研究所 上級研究員
2013年 理化学研究所 チームリーダー
2021年 横浜市立大学大学院客員教授を兼任
<メッセージ>
遺伝子発現の仕組みやクロマチン内での制御のメカニズム、ウイルスの複製や宿主との相互作用に興味のある意欲ある学生の研究室への参加を期待します。
1997年 東京大学大学院理学系研究科修了(博士(理学))
1997年 日本学術振興会 特別研究員PD
1998年 理化学研究所 協力研究員
1999年 理化学研究所 基礎科学特別研究員
2001年 理化学研究所 研究員
2004年 東京大学大学院理学系研究科 講師
2010年 東京大学大学院理学系研究科 特任准教授
2012年 理化学研究所 上級研究員
2013年 理化学研究所 チームリーダー
2021年 横浜市立大学大学院客員教授を兼任
<メッセージ>
遺伝子発現の仕組みやクロマチン内での制御のメカニズム、ウイルスの複製や宿主との相互作用に興味のある意欲ある学生の研究室への参加を期待します。
研究内容
白水グループ
細胞が何らかの刺激を受けとると、その刺激特異的にさまざまな因子が、翻訳後修飾や局在、相互作用様式を変化させ、細胞骨格や核内の転写制御因子にシグナルを伝えることで、増殖・分化などが引き起こされます。シグナルを受けとり活性化されたタンパク質は、その立体構造を変化させ、刺激がないときとは異なる因子と複合体を形成するなどダイナミックに様相を変えます。そして役割を終えたタンパク質は、不活性化され、また元の状態に戻ります。私たちは、リン酸化酵素(キナーゼ)や低分子量Gタンパク質など、シグナルにより大きく活性・状態が変化する因子を中心にして、どのように細胞内でシグナルが伝達・制御されていくのかを明らかにしようとしています。また、これらの因子は、もともとがん遺伝子として見つかった経緯をもつものが多いことからもわかるように、細胞内シグナル伝達・制御の異常は、さまざまな病気の発症につながるため、その制御機構の解明は、創薬を進める上でも非常に重要なものでもあります。
*詳しくは研究室のホームページをご覧ください(https://www.bdr.riken.jp/jp/research/labs/shirouzu-m-protein/index.html)。
*詳しくは研究室のホームページをご覧ください(https://www.bdr.riken.jp/jp/research/labs/shirouzu-m-protein/index.html)。
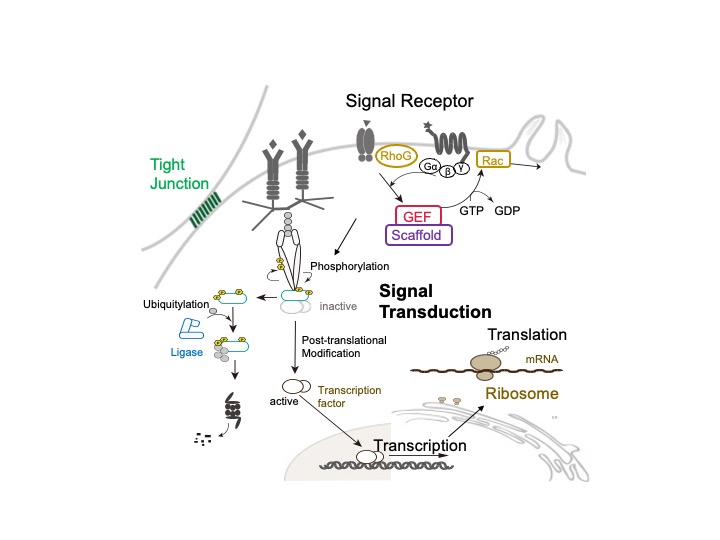 図1. 細胞内シグナル伝達・制御
図1. 細胞内シグナル伝達・制御
関根グループ
1.細胞内の転写と制御のメカニズム
真核生物の核内では、DNAはヌクレオソームに巻き取られ、高度に折りたたまれたクロマチン構造をとって収納されています。遺伝子発現のために、RNAポリメラーゼは、クロマチン内でDNAの塩基配列を読み取ってRNAを合成する必要がありますが、それをどのように行っているかは現在でもよくわかっていません。また、クロマチン内での転写制御は生物の発生やがん等の疾患とも深いつながりがあります。私たちは、RNAポリメラーゼが転写関連因子やクロマチン関連因子と形成する巨大な複合体の構造を解析し、ゲノムの転写や制御のメカニズムを解き明かすとともに、その破綻による疾患の仕組みの理解につなげます。
2. ウイルスのゲノム複製・転写のメカニズム
ウイルスが持つ固有の複製・転写装置は創薬のターゲットとなり得ます。例えば、新型コロナウイルス感染症に対して用いられるレムデシビルはウイルスのRNAポリメラーゼの働きを阻害することでウイルスの増殖を抑えるものです。私たちは、いくつかのウイルスの持つRNAポリメラーゼの構造を調べ、ウイルスが増殖する仕組みおよび宿主との相互作用の詳細を明らかにすることで、創薬研究の基礎を提供します。
*詳しくは研究室のホームページをご覧ください(https://www.bdr.riken.jp/jp/research/labs/sekine-s/index.html)。
真核生物の核内では、DNAはヌクレオソームに巻き取られ、高度に折りたたまれたクロマチン構造をとって収納されています。遺伝子発現のために、RNAポリメラーゼは、クロマチン内でDNAの塩基配列を読み取ってRNAを合成する必要がありますが、それをどのように行っているかは現在でもよくわかっていません。また、クロマチン内での転写制御は生物の発生やがん等の疾患とも深いつながりがあります。私たちは、RNAポリメラーゼが転写関連因子やクロマチン関連因子と形成する巨大な複合体の構造を解析し、ゲノムの転写や制御のメカニズムを解き明かすとともに、その破綻による疾患の仕組みの理解につなげます。
2. ウイルスのゲノム複製・転写のメカニズム
ウイルスが持つ固有の複製・転写装置は創薬のターゲットとなり得ます。例えば、新型コロナウイルス感染症に対して用いられるレムデシビルはウイルスのRNAポリメラーゼの働きを阻害することでウイルスの増殖を抑えるものです。私たちは、いくつかのウイルスの持つRNAポリメラーゼの構造を調べ、ウイルスが増殖する仕組みおよび宿主との相互作用の詳細を明らかにすることで、創薬研究の基礎を提供します。
*詳しくは研究室のホームページをご覧ください(https://www.bdr.riken.jp/jp/research/labs/sekine-s/index.html)。
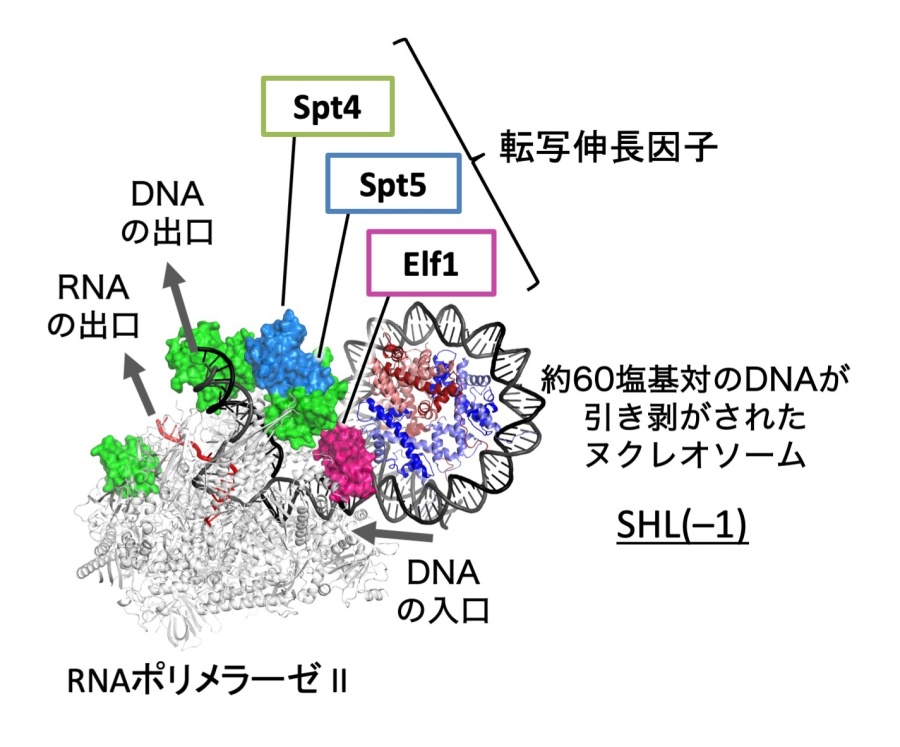 図2. RNAP II•ヌクレオソーム複合体の クライオ電子顕微鏡構造 (Science 2019)
図2. RNAP II•ヌクレオソーム複合体の クライオ電子顕微鏡構造 (Science 2019)
主要文献(Selected Publications) (*Co-first, †Correspondence)
‣Murayama K, Kato-Murayama M, Itoh Y, Miyazono K, Miyazawa K, Shirouzu M. (2020) Structural basis for inhibitory effects of Smad7 on TGF-β family signaling. J Struct Biol. 212, 107661.
‣Shoji S, Hanada K, Takahashi M, Watanabe K, Yonemochi M, Tomabechi Y, Shirouzu M. (2020) The NF-κB regulator IκBβ exhibits different molecular interactivity and phosphorylation status from IκBα in an IKK2-catalysed reaction. FEBS Lett. 594, 1532-1549. Kukimoto-Niino M, ‣Tsuda K, Ihara K, Mishima-Tsumagari C, Honda K, Ohsawa N, Shirouzu M. (2019) Structural Basis for the Dual Substrate Specificity of DOCK7 Guanine ‣Nucleotide Exchange Factor. Structure 27, 741-748.
‣Shimizu, H.,* Saito, A.,* Mikuni, J., Nakayama, E.E., Koyama, H., Honma, T., Shirouzu, M., Sekine, S.† and Shioda, T.† (2019) Discovery of a small molecule inhibitor targeting dengue virus NS5 RNA-dependent RNA polymerase. PLoS Negl. Trop. Dis. 13, e0007894.
‣Ehara, H.,* Kujirai, T.,* Fujino, Y., Shirouzu, M., Kurumizaka, H.† and Sekine, S.† (2019) Structural insight into nucleosome transcription by RNA polymerase II with elongation factors. Science 363, 744-747.
‣Kujirai, T.,* Ehara, H.,* Fujino, Y., Shirouzu, M., Sekine, S.† and Kurumizaka, H.† (2018) Structural basis of the nucleosome transition during RNA polymerase II passage. Science 362, 595-598. (†correspondence)
‣Matsuda T, Ito T, Takemoto C, Katsura K, Ikeda M, Wakiyama M, Kukimoto-Niino M, Yokoyama S, Kurosawa Y, Shirouzu M. (2018) Cell-free synthesis of functional antibody fragments to provide a structural basis for antibody-antigen interaction. PLoS One.13, e0193158.
‣Ehara, H., Yokoyama, T., Shigematsu, H., Yokoyama, S., Shirouzu, M. and Sekine, S.† (2017) Structure of the complete elongation complex of RNA polymerase II with basal factors. Science 357, 921-924. (†correspondence)
‣Sekine, S.,*† Murayama, Y.,* Svetlov, V., Nudler, E. and Yokoyama, S.† (2015) The ratcheted and ratchetable structural states of RNA polymerase underlie multiple transcriptional functions. Mol. Cell 57, 408-421
‣Shoji S, Hanada K, Takahashi M, Watanabe K, Yonemochi M, Tomabechi Y, Shirouzu M. (2020) The NF-κB regulator IκBβ exhibits different molecular interactivity and phosphorylation status from IκBα in an IKK2-catalysed reaction. FEBS Lett. 594, 1532-1549. Kukimoto-Niino M, ‣Tsuda K, Ihara K, Mishima-Tsumagari C, Honda K, Ohsawa N, Shirouzu M. (2019) Structural Basis for the Dual Substrate Specificity of DOCK7 Guanine ‣Nucleotide Exchange Factor. Structure 27, 741-748.
‣Shimizu, H.,* Saito, A.,* Mikuni, J., Nakayama, E.E., Koyama, H., Honma, T., Shirouzu, M., Sekine, S.† and Shioda, T.† (2019) Discovery of a small molecule inhibitor targeting dengue virus NS5 RNA-dependent RNA polymerase. PLoS Negl. Trop. Dis. 13, e0007894.
‣Ehara, H.,* Kujirai, T.,* Fujino, Y., Shirouzu, M., Kurumizaka, H.† and Sekine, S.† (2019) Structural insight into nucleosome transcription by RNA polymerase II with elongation factors. Science 363, 744-747.
‣Kujirai, T.,* Ehara, H.,* Fujino, Y., Shirouzu, M., Sekine, S.† and Kurumizaka, H.† (2018) Structural basis of the nucleosome transition during RNA polymerase II passage. Science 362, 595-598. (†correspondence)
‣Matsuda T, Ito T, Takemoto C, Katsura K, Ikeda M, Wakiyama M, Kukimoto-Niino M, Yokoyama S, Kurosawa Y, Shirouzu M. (2018) Cell-free synthesis of functional antibody fragments to provide a structural basis for antibody-antigen interaction. PLoS One.13, e0193158.
‣Ehara, H., Yokoyama, T., Shigematsu, H., Yokoyama, S., Shirouzu, M. and Sekine, S.† (2017) Structure of the complete elongation complex of RNA polymerase II with basal factors. Science 357, 921-924. (†correspondence)
‣Sekine, S.,*† Murayama, Y.,* Svetlov, V., Nudler, E. and Yokoyama, S.† (2015) The ratcheted and ratchetable structural states of RNA polymerase underlie multiple transcriptional functions. Mol. Cell 57, 408-421