代謝エピゲノム科学研究室
概要
様々な環境因子に晒されながらも生体が恒常性を維持できる仕組みについて、さらにそれらの破綻が疾患にいたる過程について、エピゲノム、細胞生物学、免疫学、メタボロミクス、ケミカルバイオロジーなどを組み合わせた領域横断的な研究をしています。生命現象や疾患をエピゲノム制御の観点から理解し、炎症を基盤病態とする疾患の新しい治療法の開発を目指します。
スタッフ
大学院客員教授 眞貝 洋一(しんかい よういち) ResearchMap →

<略歴>
順天堂大学大学院医学系研究科博士課程修了(1990年)医学博士。1998年京都大学ウイルス研究所助教授、2003年同教授、2011年から理化学研究所眞貝細胞記憶研究室主任研究員。
<メッセージ>
当研究室では、エピジェネティクス制御の観点から生命現象を理解することを目標に研究を行っています。このような研究を行うことで、健康とは何か、病気はどうして起きるのか、といったことの理解に繋げ、さらに病気を予防・診断したり治療したりする手だてに貢献できないかとも考え、研究しています。
順天堂大学大学院医学系研究科博士課程修了(1990年)医学博士。1998年京都大学ウイルス研究所助教授、2003年同教授、2011年から理化学研究所眞貝細胞記憶研究室主任研究員。
<メッセージ>
当研究室では、エピジェネティクス制御の観点から生命現象を理解することを目標に研究を行っています。このような研究を行うことで、健康とは何か、病気はどうして起きるのか、といったことの理解に繋げ、さらに病気を予防・診断したり治療したりする手だてに貢献できないかとも考え、研究しています。
大学院客員教授 有田 誠 (ありた まこと) ResearchMap →

<略歴>
東京大学大学院薬学系研究科博士課程修了(1997年)薬学博士。2007年から東京大学大学院薬学系研究科准教授。2014年から理化学研究所統合生命医科学研究センター(IMS)メタボローム研究チームリーダー(2018年から生命医科学研究センター)。2016年から慶應義塾大学薬学部教授を兼任。
<メッセージ>
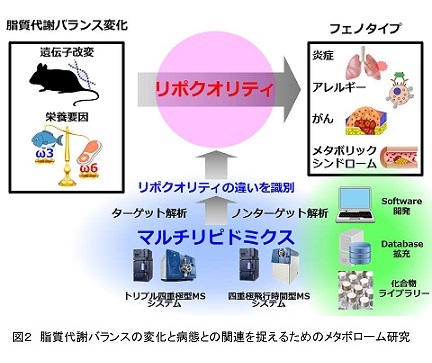
生体内には多くの種類の脂肪酸が存在し、その質の違いや代謝バランスの変化が、様々な炎症・代謝性疾患の背後に潜む重要な要素であることが示唆されています。私たちは、脂質代謝バランスと病態との関連を捉えるためのメタボローム解析を中心に、炎症を制御する創薬標的分子の同定を目指しています。
東京大学大学院薬学系研究科博士課程修了(1997年)薬学博士。2007年から東京大学大学院薬学系研究科准教授。2014年から理化学研究所統合生命医科学研究センター(IMS)メタボローム研究チームリーダー(2018年から生命医科学研究センター)。2016年から慶應義塾大学薬学部教授を兼任。
<メッセージ>
生体内には多くの種類の脂肪酸が存在し、その質の違いや代謝バランスの変化が、様々な炎症・代謝性疾患の背後に潜む重要な要素であることが示唆されています。私たちは、脂質代謝バランスと病態との関連を捉えるためのメタボローム解析を中心に、炎症を制御する創薬標的分子の同定を目指しています。
大学院客員助教 磯部 洋輔(いそべ ようすけ)

<略歴>
東京大学大学院薬学系研究科修士課程修了(2009年)薬学博士。2014年より理化学研究所生命医科学研究センター(IMS)特別研究員。2015年より同基礎科学特別研究員。2018年2月よりカリフォルニア大学バークレー校博士研究員。2020年4月より理化学研究所IMSメタボローム研究チーム研究員。2021年10月より同副チームリーダー。
<メッセージ>
生体内の脂質は炎症刺激等により酸化・代謝され、その一部は細胞内タンパク質を修飾することが知られていますが、その標的や機能的役割はほとんど未解明です。私たちは、タンパク質の修飾状態を捉えるケミカルプローブとプロテオミクスとを組み合わせた独自の手法を用い、脂質修飾の新たな標的と機能を開拓しています。さらに、修飾を模倣する化合物の探索により、脂質修飾に立脚した新たな創薬展開を目指しています。
東京大学大学院薬学系研究科修士課程修了(2009年)薬学博士。2014年より理化学研究所生命医科学研究センター(IMS)特別研究員。2015年より同基礎科学特別研究員。2018年2月よりカリフォルニア大学バークレー校博士研究員。2020年4月より理化学研究所IMSメタボローム研究チーム研究員。2021年10月より同副チームリーダー。
<メッセージ>
生体内の脂質は炎症刺激等により酸化・代謝され、その一部は細胞内タンパク質を修飾することが知られていますが、その標的や機能的役割はほとんど未解明です。私たちは、タンパク質の修飾状態を捉えるケミカルプローブとプロテオミクスとを組み合わせた独自の手法を用い、脂質修飾の新たな標的と機能を開拓しています。さらに、修飾を模倣する化合物の探索により、脂質修飾に立脚した新たな創薬展開を目指しています。
研究内容
21世紀の生命科学は、これまでの個別の分子に着目した生物学から、網羅性の高いオミックス情報を使った相互作用ネットワークの解析を通して、様々な環境因子やストレスの中で生体恒常性が維持される仕組みを理解しようとする総合科学へとシフトしています。また、メタボロミクスやケミカルバイオロジーなどの新しい技術の進歩により、生体恒常性の維持機構について深く理解するだけでなく、恒常性の破綻に起因する各種疾患の病態解明、および新しいバイオマーカーや治療法の開発を目指す研究の重要性が高まっています。
当研究室では、生体恒常性の維持機構として、一旦生じた炎症が適切に収束するための分子メカニズムについて、とくに脂質代謝系による制御機構についての研究を行っています。これまでに、アラキドン酸やEPA, DHAなどに由来する脂質メディエーターに炎症を正や負に制御する活性が見いだされており、これらの活性代謝物がいつ、どこで、どれだけ生成しているのかを包括的に捉えるための高感度一斉定量分析システムを確立しています。このような背景のもと、様々なバイオロジーや病態の背後に潜む分子メカニズムを、脂肪酸代謝バランスの変化、および活性代謝物による細胞機能制御の観点から明らかにしたいと考えています。
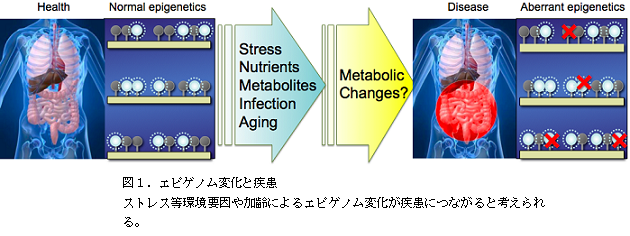
遺伝子発現のエピゲノム記憶は、遺伝子そのものの変異ではないので、加齢やストレスなどの要因によって変化しうるものです。エピゲノム調節の分子機構を理解し、また、エピゲノム調節に関わる化学修飾のネットワークによって、多様な生命機能が制御される原理を理解し、その制御が不全になることで様々な疾患が惹起されることを解明することで、エピゲノム制御の観点からの治療法の開発や創薬につなげるための基礎研究を行っています。
当研究室では、生体恒常性の維持機構として、一旦生じた炎症が適切に収束するための分子メカニズムについて、とくに脂質代謝系による制御機構についての研究を行っています。これまでに、アラキドン酸やEPA, DHAなどに由来する脂質メディエーターに炎症を正や負に制御する活性が見いだされており、これらの活性代謝物がいつ、どこで、どれだけ生成しているのかを包括的に捉えるための高感度一斉定量分析システムを確立しています。このような背景のもと、様々なバイオロジーや病態の背後に潜む分子メカニズムを、脂肪酸代謝バランスの変化、および活性代謝物による細胞機能制御の観点から明らかにしたいと考えています。
遺伝子発現のエピゲノム記憶は、遺伝子そのものの変異ではないので、加齢やストレスなどの要因によって変化しうるものです。エピゲノム調節の分子機構を理解し、また、エピゲノム調節に関わる化学修飾のネットワークによって、多様な生命機能が制御される原理を理解し、その制御が不全になることで様々な疾患が惹起されることを解明することで、エピゲノム制御の観点からの治療法の開発や創薬につなげるための基礎研究を行っています。
主要文献(Selected Publications)
‣ Tsugawa H, Ishihara T, Ogasa K, Iwanami S, Hori A, Takahashi M, Yamada Y, Satoh-Takayama N, Ohno H, Minoda A, Arita M. A lipidome landscape of aging in mice. Nature Aging (2024)
‣ Kuroha S, Katada Y, Isobe Y, Uchino H, Shishikura K, Nirasawa T, Tsubota K, Negishi K, Kurihara T, Arita M. Long chain acyl-CoA synthetase 6 facilitates the local distribution of di-docosahexaenoic acid- and ultra-long-chain-PUFA-containing phospholipids in the retina to support normal visual function in mice. FASEB Journal 37, e23151 (2023)
‣ Tsugawa H, Ikeda K, Takahashi M, Satoh A, Mori Y, Uchino H, Okahashi N, Yamada Y, Tada I, Bonini P, Higashi Y, Okazaki Y, Zhou Z, Zhu Z, Koelmel J, Cajka T, Fiehn O, Saito K, Arita M, Arita M. A lipidome atlas in MS-DIAL 4. Nature Biotechnology 38, 1159-1163 (2020)
‣ Fukuda K*. Shimura C. Miura H. Tanigawa A. Suzuki T. Dohmae D. Hiratani I. Shinkai Y*. Regulation of mammalian 3D genome organization and histone H3K9 dimethylation by H3K9 methyltransferases. Communications Biology 4(1):571, (2021)
‣ Yamada A#. Hirasawa T#. Nishimura K. Shimura C. Kogo N. Fukuda K. Kato M. Yokomori M. Hayashi T. Umeda M. Yoshimura M. Iwakura Y. Nikaido I. Itohara S. Shinkai Y*. Derepression of inflammation-related genes link to microglia activation and neural maturation defect in a mouse model of Kleefstra Syndrome. iScience 24(7):102741, (2021)
‣ Davydova E#. Shimazu T#…. Jeltsch A*. Shinkai Y*. Falnes P.Ø*. The methyltransferase METTL9 mediates pervasive 1-methylhistidine modification in mammalian proteomes. Nat Commun 12(1), 891 (2021)
‣ Kuroha S, Katada Y, Isobe Y, Uchino H, Shishikura K, Nirasawa T, Tsubota K, Negishi K, Kurihara T, Arita M. Long chain acyl-CoA synthetase 6 facilitates the local distribution of di-docosahexaenoic acid- and ultra-long-chain-PUFA-containing phospholipids in the retina to support normal visual function in mice. FASEB Journal 37, e23151 (2023)
‣ Tsugawa H, Ikeda K, Takahashi M, Satoh A, Mori Y, Uchino H, Okahashi N, Yamada Y, Tada I, Bonini P, Higashi Y, Okazaki Y, Zhou Z, Zhu Z, Koelmel J, Cajka T, Fiehn O, Saito K, Arita M, Arita M. A lipidome atlas in MS-DIAL 4. Nature Biotechnology 38, 1159-1163 (2020)
‣ Fukuda K*. Shimura C. Miura H. Tanigawa A. Suzuki T. Dohmae D. Hiratani I. Shinkai Y*. Regulation of mammalian 3D genome organization and histone H3K9 dimethylation by H3K9 methyltransferases. Communications Biology 4(1):571, (2021)
‣ Yamada A#. Hirasawa T#. Nishimura K. Shimura C. Kogo N. Fukuda K. Kato M. Yokomori M. Hayashi T. Umeda M. Yoshimura M. Iwakura Y. Nikaido I. Itohara S. Shinkai Y*. Derepression of inflammation-related genes link to microglia activation and neural maturation defect in a mouse model of Kleefstra Syndrome. iScience 24(7):102741, (2021)
‣ Davydova E#. Shimazu T#…. Jeltsch A*. Shinkai Y*. Falnes P.Ø*. The methyltransferase METTL9 mediates pervasive 1-methylhistidine modification in mammalian proteomes. Nat Commun 12(1), 891 (2021)