生命情報科学研究室
概要
タンパク質、核酸、その複合体などの立体構造形成や機能発現のメカニズムを原子レベルで明らかにするために、スーパーコンピュータを用いた分子シミュレーションによる研究や、生命医科学分野に人工知能関連技術を適用する研究を行っています。さらに、それらの方法をドラッグデザインに応用する計算創薬研究を行っています。

<略歴>
東京大学大学院農学系研究科応用生命工学専攻博士課程修了(1994年)博士(農学)。1996年から東京大学大学院農学生命科学研究科助手。2001年4月から横浜市立大学大学院助教授。2007年より同大学院准教授。2015年より同大学院教授。
<メッセージ>
タンパク質の機能発現やリガンド結合のメカニズムを分子動力学シミュレーションを主に用いて研究を進めています。製薬企業との連携やスーパーコンピュータ ポスト「京」のプロジェクトにおいて、計算創薬研究を推進しています。
東京大学大学院農学系研究科応用生命工学専攻博士課程修了(1994年)博士(農学)。1996年から東京大学大学院農学生命科学研究科助手。2001年4月から横浜市立大学大学院助教授。2007年より同大学院准教授。2015年より同大学院教授。
<メッセージ>
タンパク質の機能発現やリガンド結合のメカニズムを分子動力学シミュレーションを主に用いて研究を進めています。製薬企業との連携やスーパーコンピュータ ポスト「京」のプロジェクトにおいて、計算創薬研究を推進しています。
准教授 寺山 慧 (てらやま けい) ResearchMap →

<略歴>
京都大学大学院人間・環境学研究科共生人間学専攻博士課程修了(2016年)博士(人間・環境学)。2016年4月から東京大学大学新領域創成科学研究科特任研究員、2018年4月から理化学研究所革新知能統合研究センター特別研究員および京都大学大学院医学研究科特定助教、2018年6月から理化学研究所メディカルイノベーションハブ特別研究員(兼任)。2020年4月より横浜市立大学大学院准教授。
<メッセージ>
機械学習・最適化・コンピュータビジョン等情報科学の手法とそれらを創薬・材料科学・化学・水産業・海洋工学等の現実的な課題に応用する研究を行なっています。特に、分子動力学シミュレーションや量子化学シミュレーションと強化学習・深層学習を組み合わせることで創薬を加速したりタンパク質の性質を解析する研究を進めています。
京都大学大学院人間・環境学研究科共生人間学専攻博士課程修了(2016年)博士(人間・環境学)。2016年4月から東京大学大学新領域創成科学研究科特任研究員、2018年4月から理化学研究所革新知能統合研究センター特別研究員および京都大学大学院医学研究科特定助教、2018年6月から理化学研究所メディカルイノベーションハブ特別研究員(兼任)。2020年4月より横浜市立大学大学院准教授。
<メッセージ>
機械学習・最適化・コンピュータビジョン等情報科学の手法とそれらを創薬・材料科学・化学・水産業・海洋工学等の現実的な課題に応用する研究を行なっています。特に、分子動力学シミュレーションや量子化学シミュレーションと強化学習・深層学習を組み合わせることで創薬を加速したりタンパク質の性質を解析する研究を進めています。
助教 浴本 亨(えきもと とおる) ResearchMap →

<略歴>
九州大学大学院理学府物理学専攻博士課程修了(2011年)博士(理学)。2011年4月から京都大学化学研究所博士研究員、2013年4月より横浜市立大学大学院博士研究員、特任助教。2018年4月より同大学院助教。
<メッセージ>
疾患や感染病関連タンパク質の機能発現メカニズムを、分子動力学シミュレーションを用いて研究しています。タンパク質の動的構造変化を、スーパーコンピュータによる大規模計算や実験と連携した手法により解析し、動的機能を制御する創薬へ貢献したいと考えています。
九州大学大学院理学府物理学専攻博士課程修了(2011年)博士(理学)。2011年4月から京都大学化学研究所博士研究員、2013年4月より横浜市立大学大学院博士研究員、特任助教。2018年4月より同大学院助教。
<メッセージ>
疾患や感染病関連タンパク質の機能発現メカニズムを、分子動力学シミュレーションを用いて研究しています。タンパク質の動的構造変化を、スーパーコンピュータによる大規模計算や実験と連携した手法により解析し、動的機能を制御する創薬へ貢献したいと考えています。
研究内容
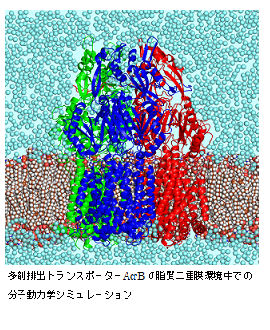
コンピュータの急速な性能向上を背景として、生命科学のデータに対してコンピュータで高度に情報処理を行う研究が発展しています。タンパク質や核酸(DNA, RNA)といった生体高分子の働き(機能)は、その原子レベルの立体構造を基盤として、分子の運動として理解可能です。そこで、スーパーコンピュータなどの高速なコンピュータを活用して、分子動力学シミュレーションなどの計算的方法によって、生体高分子の機能発現メカニズムを分子レベルで解明することを目指しています。例えば、分子シミュレーションのターゲットとして注目しているものに膜タンパク質があります。多くの医薬品の標的タンパク質も膜タンパク質です。これまでの成果のひとつとして、左下図に示した多剤排出トランスポーターAcrBの研究があります。多剤排出トランスポーターは、薬が効かなくなる薬剤耐性の原因でもあり、その薬剤排出メカニズムの解明が重要とされています。さらに、製薬企業とも連携しつつ、分子シミュレーションを創薬に応用するin silicoドラッグデザイン研究も展開しています。
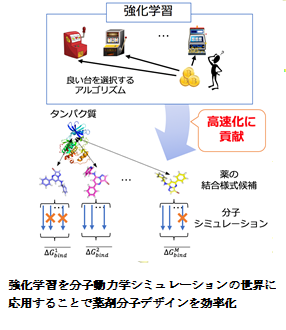
また、近年人工知能関連技術が注目されており、顔認証や機械翻訳など様々な社会実装が進みつつあります。本研究室では、人工知能の基盤となる機械学習や大域最適化手法等を用いてシミュレーションの高速化・高精度化や新しい薬剤候補になる化合物の設計にも取り組んでいます。これまでの成果の一つとして、強化学習と呼ばれる機械学習の一手法を分子シミュレーションの世界に応用することで、薬剤候補分子とタンパク質の結合様式を高速かつ高精度に推定する研究があります。薬剤候補分子とタンパク質の結合様式は薬を設計する際に不可欠な情報です。しかし、多数の結合様式の候補から分子シミュレーションで実際の結合様式を見極めるためには莫大な計算コストがかかります。ここで結合様式を絞り込む状況を強化学習の枠組みで捉え、最適なアルゴリズムを適用することで計算コストを半分から5分の1程度に削減することができます。このように、生命科学の様々な場面に機械学習や最適化手法を応用しすることで、研究を加速する・新たな発見を助ける手法開発を進めています。
また、近年人工知能関連技術が注目されており、顔認証や機械翻訳など様々な社会実装が進みつつあります。本研究室では、人工知能の基盤となる機械学習や大域最適化手法等を用いてシミュレーションの高速化・高精度化や新しい薬剤候補になる化合物の設計にも取り組んでいます。これまでの成果の一つとして、強化学習と呼ばれる機械学習の一手法を分子シミュレーションの世界に応用することで、薬剤候補分子とタンパク質の結合様式を高速かつ高精度に推定する研究があります。薬剤候補分子とタンパク質の結合様式は薬を設計する際に不可欠な情報です。しかし、多数の結合様式の候補から分子シミュレーションで実際の結合様式を見極めるためには莫大な計算コストがかかります。ここで結合様式を絞り込む状況を強化学習の枠組みで捉え、最適なアルゴリズムを適用することで計算コストを半分から5分の1程度に削減することができます。このように、生命科学の様々な場面に機械学習や最適化手法を応用しすることで、研究を加速する・新たな発見を助ける手法開発を進めています。
主要文献(Selected Publications)
‣S. Tojo, Z. Zhang, H. Matsui, M. Tahara, M. Ikeguchi, M. Kochi, M. Kamada, H. Shigematsu, A. Tsutsumi, N. Adachi, T. Shibata, M. Yamamoto, M. Kikkawa, T. Senda, Y. Isobe, U. Ohto, T. Shimizu: Structural analysis reveals TLR7 dynamics underlying antagonism. Nat. Commun. 11(1), 5204 (2020).
‣S. Matsumoto, S. Ishida, M. Araki, T. Kato, K. Terayama, Y. Okuno: Extraction of protein dynamics information from cryo-EM maps using deep learning, Nature Machine Intelligence, 3, 153-160, (2021).
‣N. Ienaga, K. Higuchi, T. Takashi, K. Gen, K. Tsuda, K. Terayama: Vision-based egg quality prediction in Pacific bluefin tuna (Thunnus orientalis) by deep neural network, Scientific Reports, 11, 6, (2021).
‣T. Muraoka, D. Noguchi, RS. Kasai, K. Sato, R. Sasaki, KV. Tabata, T. Ekimoto, M. Ikeguchi, K. Kamagata, N. Hoshino, H. Noji, T. Akutagawa, K. Ichimura, K. Kinbara: A synthetic ion channel with anisotropic ligand response, Nat. Commun. 11(1), 2924 (2020).
‣S. Kori, T. Jimenji, T. Ekimoto, M. Sato, F. Kusano, T. Oda, M. Unoki, M. Ikeguchi, K. Arita: Serine 298 phosphorylation in linker 2 of UHRF1 regulates ligand-binding property of its tandem tudor domain. J. Mol. Biol. 432, 4061-4075 (2020).
‣S. Matsumoto, S. Ishida, M. Araki, T. Kato, K. Terayama, Y. Okuno: Extraction of protein dynamics information from cryo-EM maps using deep learning, Nature Machine Intelligence, 3, 153-160, (2021).
‣N. Ienaga, K. Higuchi, T. Takashi, K. Gen, K. Tsuda, K. Terayama: Vision-based egg quality prediction in Pacific bluefin tuna (Thunnus orientalis) by deep neural network, Scientific Reports, 11, 6, (2021).
‣T. Muraoka, D. Noguchi, RS. Kasai, K. Sato, R. Sasaki, KV. Tabata, T. Ekimoto, M. Ikeguchi, K. Kamagata, N. Hoshino, H. Noji, T. Akutagawa, K. Ichimura, K. Kinbara: A synthetic ion channel with anisotropic ligand response, Nat. Commun. 11(1), 2924 (2020).
‣S. Kori, T. Jimenji, T. Ekimoto, M. Sato, F. Kusano, T. Oda, M. Unoki, M. Ikeguchi, K. Arita: Serine 298 phosphorylation in linker 2 of UHRF1 regulates ligand-binding property of its tandem tudor domain. J. Mol. Biol. 432, 4061-4075 (2020).