
ここでは NMR を使って、蛋白質の立体構造をどのようにして決定するのかを簡単に示します。(要約編)

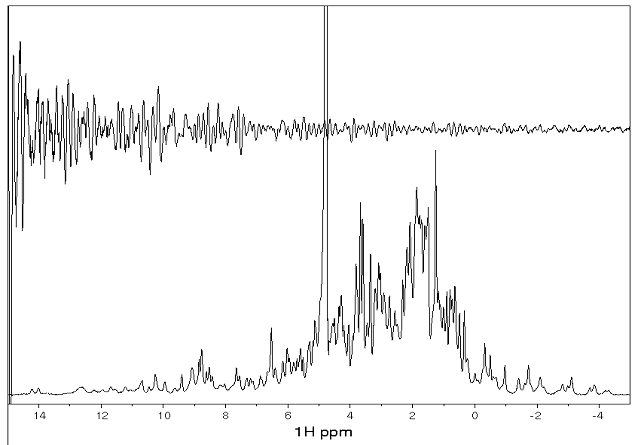
蛋白質の溶液を上図のような試料管に入れ NMR 装置にセットし電磁波を照射すると、下図のような FID と呼ばれる信号を検出することができます。これをフーリエ変換したものが、そのすぐ下に示した NMR スペクトルです。この例は一次元のスペクトルです。蛋白質に存在する各水素原子核がそれぞれ1本のピークを呈しますので、千本以上のピークが重なっており、これでは解析が大変困難です。
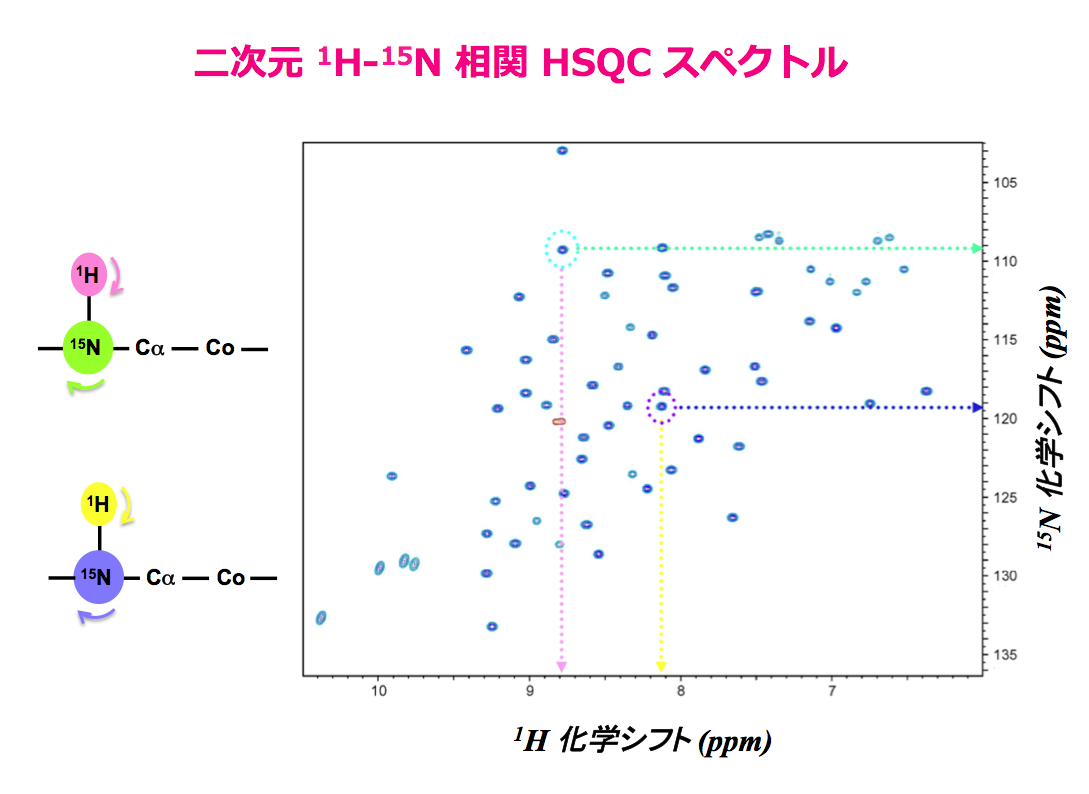
しかし、スペクトルを二次元に展開すると、ピークどうしの重なりはかなりましになります。下図は各アミノ酸のアミド基における、H と N 核の相関を示すスペクトルで、青いピークのそれぞれが蛋白質上のどれかのアミノ酸のアミド基に対応します。したがって、ピークの数はアミノ酸の数とほぼ同じです(厳密にいうと、アスパラギン Asn, グルタミン Gln, トリプトファン Trp などの側鎖にあるアミノ基由来のピークも見えていますので、その分だけピークの数は多くなります)。
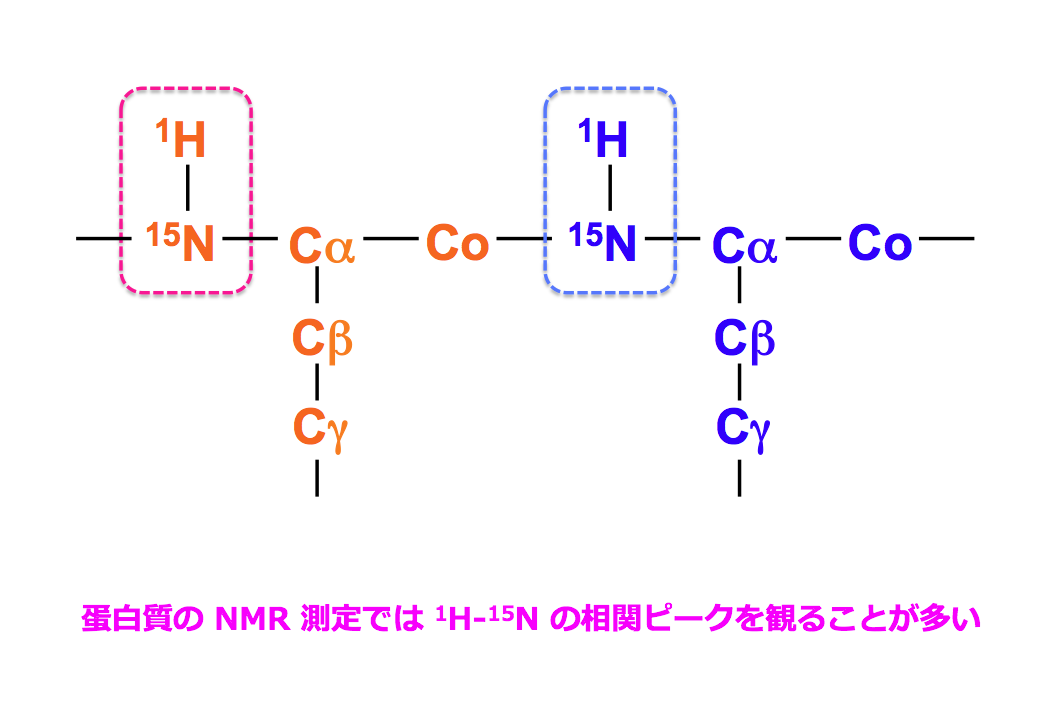
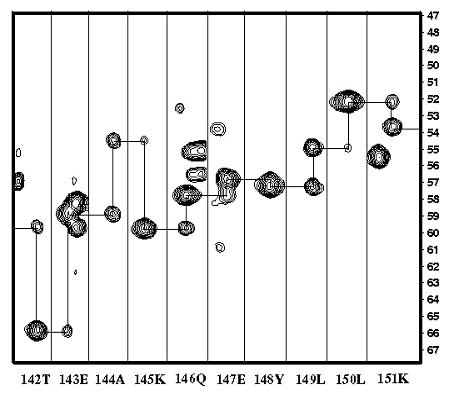
スペクトルの次元数を増やすほど分かりやすくなります。下のスペクトルは、3次元の HNCA スペクトルの一部を短冊形に切り取って、アミノ酸の配列の順番に沿って並べ直したものです。連鎖帰属と呼ばれる方法により、それぞれのピークが蛋白質上のどの原子核のペアに由来したのかを決定することが出来ます。この帰属により、各原子核のスピンが NMR の静磁場の中でどの程度のスピードで歳差運動しているのかを求めることができ、化学シフト値(単位は ppm)の形で登録されます。この化学シフト値は今後の全てのNMRの解析で必要となる、もっとも重要な基本的データとなります。
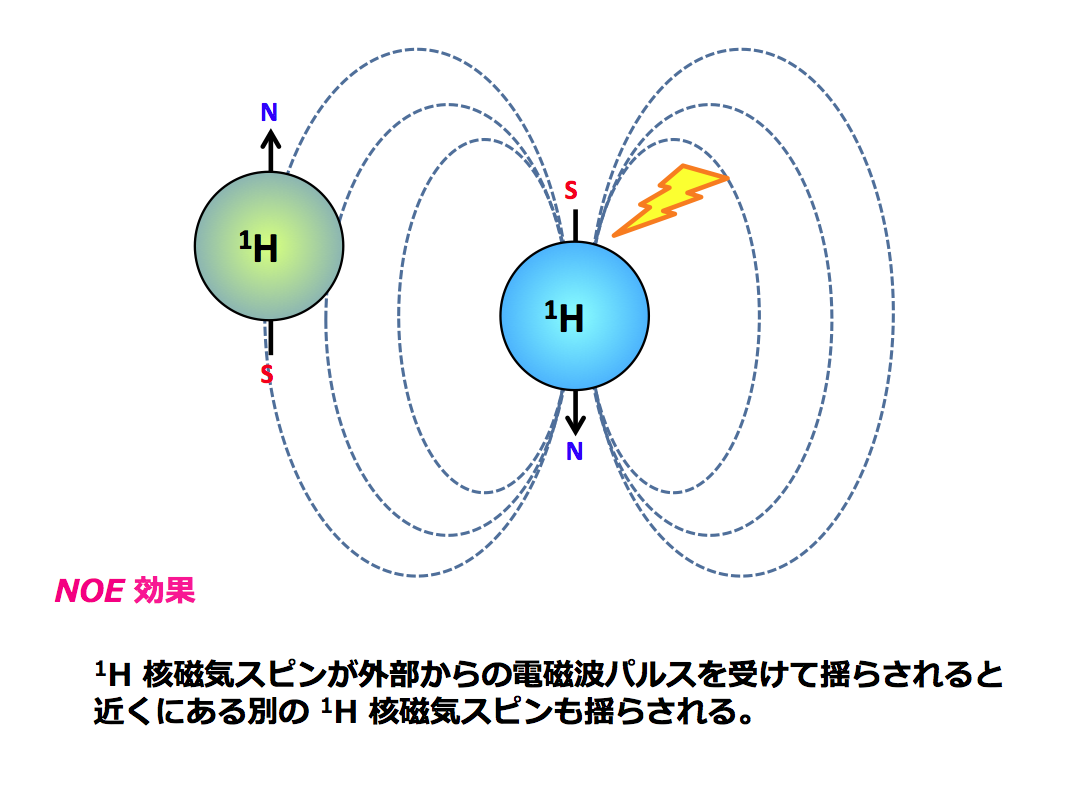
蛋白質が正常に折りたたまれていて、もし、核 A と B が空間的に近い場合、NOESY と呼ばれるスペクトルを測定すると、両者の間にピークが生じます。先ほどの帰属の結果を参照すると、横軸と縦軸の化学シフト値から、そのピークが核 A と B に対応していることが分かります。これが立体構造を決定するために重要な距離情報です。核 A と B は一次構造上では離れているかもしれません。しかし、立体構造上では近くどうしにあるという情報が重要で、これは距離制限と呼ばれています。
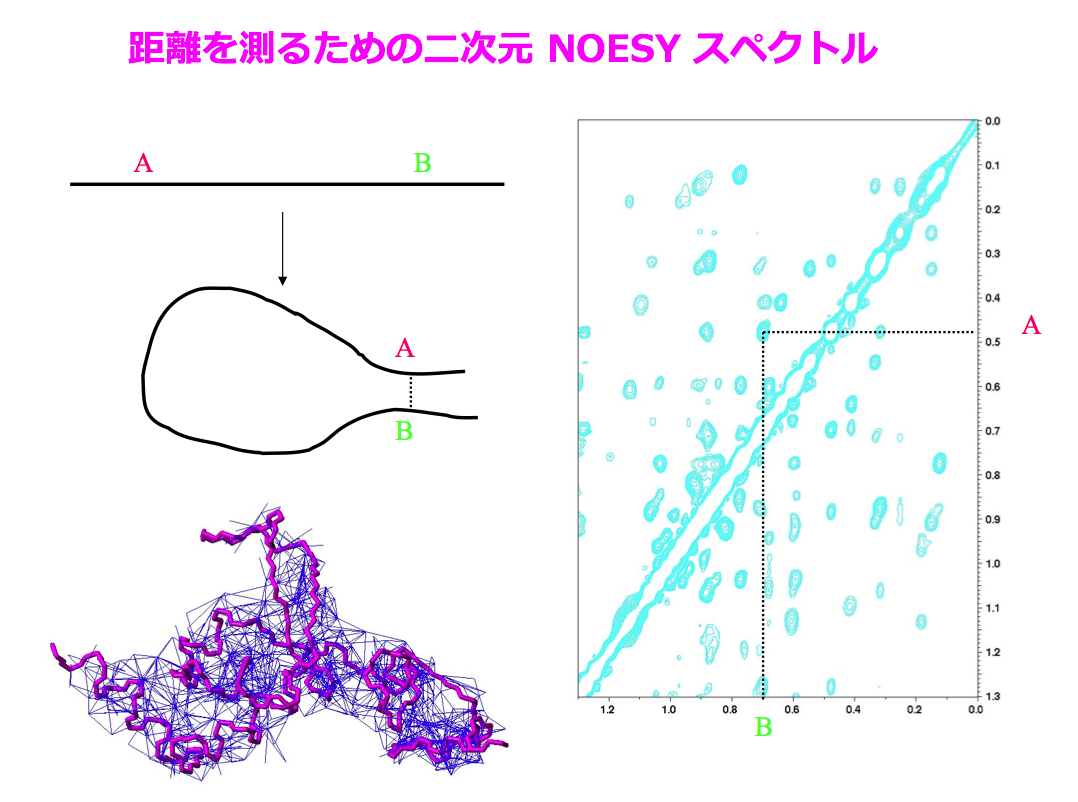
NOE ピークから得られた 1,500 個以上の距離情報はコンピュータに入力され、それら全てを満たす立体構造が試行錯誤的に決定されます。下図の短い青い線は一つ一つの距離制限を示します。残念ながら NOE は核間距離が短い(5~6オングストローム以内)場合にのみ観測されるため、多数の距離制限を集めないと正確で精密な構造が得られません。結合の間の角度(二面角)もスカラー結合定数 J という値から求めることが出来、それらを角度情報として構造の組み立てに使うことが出来ます。しかし、これも距離情報と同じく近距離に相当する制限情報であることに変わりはありません。